csvdata <- read.csv(
file = "ovary_data.csv",
header = T,
sep = ",",
stringsAsFactors = F
) 数据的读取与输出
1 基于R基础包的数据读取
1.1 读取.CSV数据文件
read.csv或read.table均可
header:第一行是否是列名sep:字段分隔符。文件每行上的值由此字符分隔。read.table的默认值为sep = “”,表示分隔符为‘空白’,即一个或多个空格、制表符、换行符或回车。read.csv的默认值为sep = ",",表示分隔符为英文逗号stringsAsFactors:是否将字符向量转换为因子
csvdata <- read.table(
"ovary_data.csv",
header = T,
sep = ",",
row.names = "patientID",
colClasses = c("character", "character", "character", "numeric", "numeric", "numeric")
) -
colClasses: (可选)指定每一列的变量类型 -
as.is:该参数用于确定read.table()函数读取字符型数据时是否转换为因子型变量。当其取值为FALSE时,该函数将把字符型数据转换为因子型数据,取值为TRUE时,仍将其保留为字符型数据。
1.2 读取.txt文件
refGene <- read.table(
"refGene.txt",
header = F,
sep = "\t"
)1.3 读取.xlsx/.xls文件
MacOS 首选 gdata 包(因自带perl语言);Windows首选 xlsx 包
1.4 读取.sav文件
.sav文件是SPSS的输出文件,可以通过foreign包的read.spss()函数来读取。
1.5 读取程序包中的案例数据集
1.6 下载和读取压缩包
1.6.1 解压.zip文件
unzip(
zipfile = "test.zip",
files = "ferroptosis_suppressor.csv",
overwrite = T
)zipfile:压缩包的位置及文件名files:(可选)需要提取的文件的文件名,默认解压压缩包内的所有文件overwrite:解压后是否覆盖同名文件
1.6.2 解压.tar文件
untar(
"test.tar", # 压缩包的位置及文件名
files = "ferroptosis_suppressor.csv" # 提取指定文件,默认解压压缩包内的所有文件
) 1.6.3 下载和解压.gz或.bz2文件
这两个压缩文件与前面的相比比较特别,因为.gz或.bz2文件,可以称之为压缩文件,也可以直接作为一个数据文件进行读取。
#下载gz文件
download.file(
"http://hgdownload.soe.ucsc.edu/goldenPath/hg38/database/refGene.txt.gz",
destfile = "refGene.txt.gz") # 目标下载位置,注意需要添加后缀名
#直接以数据的形式读取.gz文件
mydata <- read.table("refGene.txt.gz")list.files()在批量读取文件时十分有用,因为它能够获取指定目录下的文件名
list.files(
path = "folder_name",
pattern = ".docx$",
full.names = T
)path:需要列出的文件所在的路径,若忽略此项则列出当前工作路径下的所有文件pattern:列出目录中包含指定字符的文件。如pattern = "\\.docx$"表示列出所有以“.docx”为后缀的文件;pattern = "^G"列出所有文件名以“G”开头的文件full.names:FALSE:仅输出文件名;TRUE(默认):输出路径+文件名
1.7 手动生成数据框
framdata <- data.frame(
y = c(6, 8, 12, 14, 14, 15, 17, 22, 24, 23),
x1 = c(2, 5, 4, 3, 4, 6, 7, 5, 8, 9),
x2 = c(14, 12, 12, 13, 7, 8, 7, 4, 6, 5)
)
framdata y x1 x2
1 6 2 14
2 8 5 12
3 12 4 12
4 14 3 13
5 14 4 7
6 15 6 8
7 17 7 7
8 22 5 4
9 24 8 6
10 23 9 5# 使用文本编辑器直接在窗口中编辑数据。macOS需要安装XQuartz(www.xquartz.org)才能运行此代码。
framdata <- edit(framdata)
2 基于readr包的数据读取

readr包是tidyverse中用于数据读取的R包。相较于R基础包自带的数据读取函数,基于readr包的数据读取有以下优势:
一般来说,
readr比基础包中的函数要更快(快大约10-100倍);如果只要求快速读取大容量数据的话,也可以使用data.tabale包中的fread()函数。在某些情况下fread()比readr的读取速度略快,但是readr能够更好的兼容基于tidyverse的数据处理流程。readr可以生成数据框tibble,并且不会将字符向量转换为因子,不使用行名称,也不会随意改动列名称。这些都是R基础包使用中令人沮丧的事情。readr更易于重复使用。R基础包则需要继承操作系统的功能,并依赖环境变量,同样的代码在另一个电脑上不一定能正常运行。
2.1 读取.csv文件
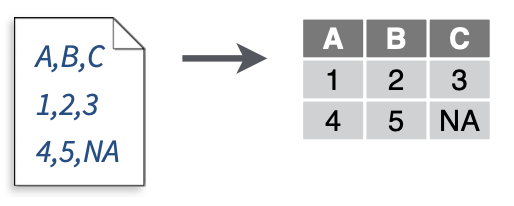
这里我们使用students.csv案例数据,该数据可以从此处下载。对于.csv文件,我们可以通过read_csv()读取:
students <- read_csv("data/r_basic/students.csv")# 也可以直接从URL读取
students <- read_csv("https://pos.it/r4ds-students-csv")当运行 read_csv() 时,它会打印出一条消息,报告数据的行数和列数、使用的分隔符和列类型。int 代表整数型数据,dbl 代表数值型数据,chr 代表字符型数据,dttm 代表日期时间型数据。这些变量非常重要,因为对列进行的操作在很大程度上取决于该列的 “类型”。它还告诉我们可以通过spec()来提取所有列的类型信息。
students# A tibble: 6 × 6
...1 `Student ID` `Full Name` favourite.food mealPlan AGE
<dbl> <dbl> <chr> <chr> <chr> <chr>
1 1 1 Sunil Huffmann Strawberry yoghurt Lunch only 4
2 2 2 Barclay Lynn French fries Lunch only 5
3 3 3 Jayendra Lyne N/A Breakfast and lu… 7
4 4 4 Leon Rossini Anchovies Lunch only <NA>
5 5 5 Chidiegwu Dunkel Pizza Breakfast and lu… five
6 6 6 Güvenç Attila Ice cream Lunch only 6 同时,我们注意到通过read_csv读取后的数据是一个 tibble,这是一种升级版的data.frame,被 tidyverse 用来避免一些data.frame的常见问题。Tibbles 和data.frame之间的一个重要的区别在于 tibbles 打印数据的方式:tibbles 是为大型数据集设计的,因此只显示前几行和适应屏幕宽度的列。可以使用 print(flights, width = Inf) 显示所有列,或者使用 glimpse()(见基于dplyr包的数据处理)。因为这里的数据本身只有6行6列,所以所有数据都被显示了出来。
另一个问题是,“Student ID”和“Full Name”列名被单引号扩起来。这是因为这两列的列名中包含了空格,这不符合R语言列名规范,也体现了tibble不会改变原有列名的特点。如果是用read.csv()读取的话,这两列中间的空格会被”.”替换。下面我们将这两个列名中的空格用下划线“_”替换以符合规范:
rename(
students,
student_id = `Student ID`,
full_name = `Full Name`
)# A tibble: 6 × 6
...1 student_id full_name favourite.food mealPlan AGE
<dbl> <dbl> <chr> <chr> <chr> <chr>
1 1 1 Sunil Huffmann Strawberry yoghurt Lunch only 4
2 2 2 Barclay Lynn French fries Lunch only 5
3 3 3 Jayendra Lyne N/A Breakfast and lunch 7
4 4 4 Leon Rossini Anchovies Lunch only <NA>
5 5 5 Chidiegwu Dunkel Pizza Breakfast and lunch five
6 6 6 Güvenç Attila Ice cream Lunch only 6 rename()要求新变量名在 = 的左侧,旧变量名在右侧。和其他dplyr中的函数一样,rename不会对原始数据进行修改,因此需要将rename后的数据重新赋值给新的对象或覆盖原来的对象以应用对变量名的修改(更多关于rename()的说明详见后续章节)。这里为了后续演示,没有重新赋值。
另一种方法是使用 janitor 包中的 clean_names() 函数,一次性将不符合规范的列名规范化重命名:
janitor::clean_names(students)2.1.1 定义缺失值
通过检查该数据,发现”favourite.food“一列中有一个“N/A”字符,这在原始数据的录入时表示缺失值。但是read_csv()默认将空字符串(“”)识别为缺失值NA。我们可以在读取数据时添加额外的参数让read_csv()能够将“N/A”识别为缺失值:
# A tibble: 6 × 5
`Student ID` `Full Name` favourite.food mealPlan AGE
<dbl> <chr> <chr> <chr> <chr>
1 1 Sunil Huffmann Strawberry yoghurt Lunch only 4
2 2 Barclay Lynn French fries Lunch only 5
3 3 Jayendra Lyne <NA> Breakfast and lunch 7
4 4 Leon Rossini Anchovies Lunch only NA
5 5 Chidiegwu Dunkel Pizza Breakfast and lunch five
6 6 Güvenç Attila Ice cream Lunch only 6 2.1.2 定义列类别
读入数据后的另一个常见操作是更改变量/列类型。例如,这个数据中“favourite_food”,“meal_plan”应该是因子型变量;“age”应该是数值型变量,因此我们需要对其进行转换。
这里我们使用管道符|>来简化代码,对其的详细说明参考后续章节。mutate()的作用是根据现有列计算并添加新列(见基于dplyr包的数据处理-mutate())。对于因子变量的转换,使用base包的factor(),转换后的变量仍命名为“meal_plan”,所以它会覆盖原有的“meal_plan”变量。对于数值型变量的转换,这里用readr中的parse_number()函数:
students |>
janitor::clean_names() |>
mutate(
favourite_food = factor(favourite_food),
meal_plan = factor(meal_plan),
age = parse_number(age)
)Warning: There was 1 warning in `mutate()`.
ℹ In argument: `age = parse_number(age)`.
Caused by warning:
! 1 parsing failure.
row col expected actual
5 -- a number five# A tibble: 6 × 5
student_id full_name favourite_food meal_plan age
<dbl> <chr> <fct> <fct> <dbl>
1 1 Sunil Huffmann Strawberry yoghurt Lunch only 4
2 2 Barclay Lynn French fries Lunch only 5
3 3 Jayendra Lyne <NA> Breakfast and lunch 7
4 4 Leon Rossini Anchovies Lunch only NA
5 5 Chidiegwu Dunkel Pizza Breakfast and lunch NA
6 6 Güvenç Attila Ice cream Lunch only 6⚠️注意,此时的输出结果中出现了警告信息。提示部分“age”数据出现了解析失败,具体在第五行,是一个“five”值,它不能被解析成数值,所以我们对其进行以下处理:
students <- students |>
janitor::clean_names() |>
mutate(
favourite_food = factor(favourite_food),
meal_plan = factor(meal_plan),
age = parse_number(if_else(age == "five", "5", age))
)
students# A tibble: 6 × 5
student_id full_name favourite_food meal_plan age
<dbl> <chr> <fct> <fct> <dbl>
1 1 Sunil Huffmann Strawberry yoghurt Lunch only 4
2 2 Barclay Lynn French fries Lunch only 5
3 3 Jayendra Lyne <NA> Breakfast and lunch 7
4 4 Leon Rossini Anchovies Lunch only NA
5 5 Chidiegwu Dunkel Pizza Breakfast and lunch 5
6 6 Güvenç Attila Ice cream Lunch only 6readr提供了一系列函数用于转换变量类型:
-
parse_factor():类似于base包的factor(),但是如果通过levels参数指定了因子水平,并且该变量的某些值在这些水平中找不到,则会给出警告并将这些值转换成缺失值NA。而factor()则不会给出警告信息。[1] cat dog <NA> Levels: cat dog cowparse_factor(x, levels = animals)[1] cat dog <NA> attr(,"problems") # A tibble: 1 × 4 row col expected actual <int> <int> <chr> <chr> 1 3 NA value in level set caw Levels: cat dog cow -
parse_number():转换数值型变量。这个函数会解析它找到的第一个数字,然后删除第一个数字之前的所有非数字字符和第一个数字之后的所有字符,同时也会忽略千分位分隔符“,”。parse_number("$1,000") # leading `$` and grouping character `,` ignored[1] 1000parse_number("euro1,000") # leading non-numeric euro ignored[1] 1000parse_number("t1000t1000") # only parses first number found[1] 1000parse_number("1,234.56")[1] 1234.56# explicit locale specifying European grouping and decimal marks parse_number("1.234,56", locale = locale(decimal_mark = ",", grouping_mark = "."))[1] 1234.56# SI/ISO 31-0 standard spaces for number grouping parse_number("1 234.56", locale = locale(decimal_mark = ".", grouping_mark = " "))[1] 1234.56 parse_integer():转换整数型变量。较少使用,因为整数型变量和数值型变量或称doubles型变量的本质是一样的。-
parse_datetime():转换日期/时间变量。parse_datetime("01/02/2010", "%d/%m/%Y")[1] "2010-02-01 UTC"parse_datetime("01/02/2010", "%m/%d/%Y")[1] "2010-01-02 UTC"parse_datetime("2010/01/01 12:00 US/Central", "%Y/%m/%d %H:%M %Z")[1] "2010-01-01 18:00:00 UTC"
这些变量类型转换函数都通过了缺失值定义参数,如:
# Specifying strings for NAs
parse_number(c("1", "2", "3", "NA"))[1] 1 2 3 NAparse_number(c("1", "2", "3", "NA", "Nothing"), na = c("NA", "Nothing"))[1] 1 2 3 NA NA因此,既可以像上面的定义缺失值中一样在数据读取时就定义数据集中的缺失值,也可以在转换变量类型时分别定义每一列的缺失值。
2.1.3 非标准数据的读取
通常,read_csv()使用数据的第一行作为列名,这是一种非常常见的约定。但在有时候数据的顶部可能包含几行元数据。这时候可以通过添加skip参数让read_csv()跳过前n行,也可以通过comment参数让其跳过以特定字符(例如“#”)开头的所有行。
read_csv(
"The first line of metadata
The second line of metadata
x,y,z
1,2,3",
skip = 2
)# A tibble: 1 × 3
x y z
<dbl> <dbl> <dbl>
1 1 2 3read_csv(
"# A comment I want to skip
x,y,z
1,2,3",
comment = "#" # 跳过以“#”开头的行
)# A tibble: 1 × 3
x y z
<dbl> <dbl> <dbl>
1 1 2 3在其他情况下,数据可能没有列名。可以使用 col_names = FALSE 告知 read_csv() 不要将第一行视为列名,而是按从 “X1” 到 “Xn” 的顺序标记它们:
read_csv(
"1,2,3
4,5,6",
col_names = FALSE
)# A tibble: 2 × 3
X1 X2 X3
<dbl> <dbl> <dbl>
1 1 2 3
2 4 5 6或者,可以用 col_names 参数以包含列名的字符向量来自定义列名:
2.2 其他类型数据文件的读取
一旦掌握了read_csv()的语法,其他数据格式的读取只需调用特定的函数即可,语法和read_csv()类似:
-
read_csv2():读取以分号“;”分隔的数据。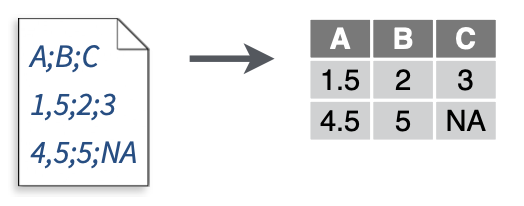
read_tsv()和read_table():读取以制表符分隔的数据。-
read_fwf():读取固定宽度数据文件,其中列以空格分隔。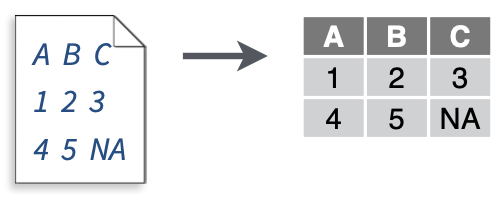
-
read_delim():读取以任意分隔符(例如“|”)分隔的数据。如果没有指定分隔符那么read_delim()会尝试自动猜测分隔符。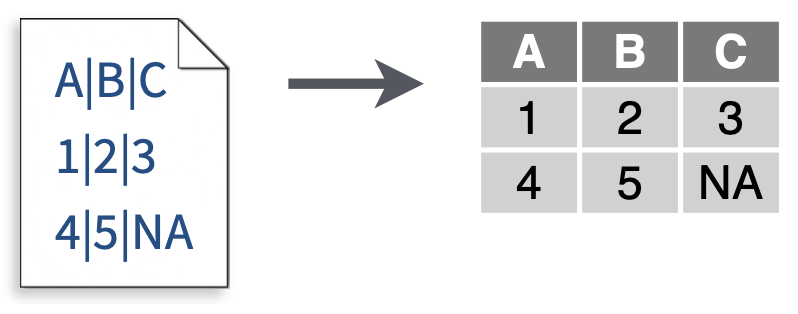
2.3 从多个文件读取数据
有时,数据会拆分到多个文件中,而不是包含在单个文件中。例如,假设这里有多个月的销售数据,每个月的数据都在一个单独的文件中,分别是: 1月的销售数据“01-sales.csv”、2月的销售数据“02-sales.csv”和三月的销售数据“03-sales.csv”。通过使用 read_csv() ,可以一次读取这些数据,并合并成一个数据。
list.files(
"data/r_basic",
pattern = "sales\\.csv$",
full.names = TRUE
) %>%
read_csv(id = "file")# A tibble: 19 × 6
file month year brand item n
<chr> <chr> <dbl> <dbl> <dbl> <dbl>
1 data/r_basic/01-sales.csv January 2019 1 1234 3
2 data/r_basic/01-sales.csv January 2019 1 8721 9
3 data/r_basic/01-sales.csv January 2019 1 1822 2
4 data/r_basic/01-sales.csv January 2019 2 3333 1
5 data/r_basic/01-sales.csv January 2019 2 2156 9
6 data/r_basic/01-sales.csv January 2019 2 3987 6
7 data/r_basic/01-sales.csv January 2019 2 3827 6
8 data/r_basic/02-sales.csv February 2019 1 1234 8
9 data/r_basic/02-sales.csv February 2019 1 8721 2
10 data/r_basic/02-sales.csv February 2019 1 1822 3
11 data/r_basic/02-sales.csv February 2019 2 3333 1
12 data/r_basic/02-sales.csv February 2019 2 2156 3
13 data/r_basic/02-sales.csv February 2019 2 3987 6
14 data/r_basic/03-sales.csv March 2019 1 1234 3
15 data/r_basic/03-sales.csv March 2019 1 3627 1
16 data/r_basic/03-sales.csv March 2019 1 8820 3
17 data/r_basic/03-sales.csv March 2019 2 7253 1
18 data/r_basic/03-sales.csv March 2019 2 8766 3
19 data/r_basic/03-sales.csv March 2019 2 8288 6这些数据可以从https://pos.it/r4ds-01-sales, https://pos.it/r4ds-02-sales, https://pos.it/r4ds-03-sales下载。下载后的数据被翻到了”data”文件夹下的“r_basic”子文件夹内。因此,我们可以通过list.files()并结合pattern参数列出我们需要的销售数据文件,pattern中应用了正则表达式,这在接下来的章节中会详细说明。read_csv中的id参数会在结果数据中添加一个新列,用于标识数据来自哪个文件。
2.4 手动生成tibble
有时需要在R脚本中“手动”输入一些数据来生成一个tibble。有两个函数可以做到这一点,这两个函数的不同之处在于是按列还是按行布局tibble。其中 tibble() 按列工作,类似于base包中的data.fram():
# A tibble: 3 × 3
x y z
<dbl> <chr> <dbl>
1 1 h 0.08
2 2 m 0.83
3 5 g 0.6 按列布局数据很难看到行之间是如何关联的,因此可以选择 tribble() ,这是“Transsposed Tibble”的缩写,它允许逐行布局数据。列标题以 ~ 开头,条目之间用逗号分隔。这使得以易于阅读的形式布局少量数据成为可能:
tribble(
~x, ~y, ~z,
1, "h", 0.08,
2, "m", 0.83,
5, "g", 0.60
)# A tibble: 3 × 3
x y z
<dbl> <chr> <dbl>
1 1 h 0.08
2 2 m 0.83
3 5 g 0.6 3 数据的导出
3.1 基于base包的数据导出
3.1.1 导出.csv文件
write.csv(
mydata,
row.names = F,#是否输出行名称
"mydata.csv"
)3.1.2 导出.xlsx/.xls文件
3.1.3 储存为.Rdata/.RDS文件
在R中保存数据的更好的方法是通过save()将数据导出为.Rdata文件或通过saveRDS()保存为.RDS文件,它们以R的定制二进制格式存储数据。这意味着当重新加载这些对象时,加载的是与当时存储时的完全相同的R对象。它们能够最大程度的保持数据的原貌,如对变量类型的定义,同时可以将图像、列表等对象导出。
3.2 基于readr包的数据导出
3.2.1 导出.csv/.txt文件
Readr提供了两个用于将数据写回磁盘的函数: write_csv() 和 write_tsv() 。这些函数最重要的参数是 x (要保存的数据集的名字)和 file (要保存的位置)。还可以使用 na 指定如何写入缺失值,以及通过 append 定义是否覆盖写入现有文件。
现在我们将之前的“students”数据导出,然后重新读取:
用这种方式导出的文件无法保留我们定义的变量类型信息。因此,更好的方式是和上面一样保存为.Rdata文件或.RDS文件。和base包的saveRDS()和readRDS()对应的,readr包中也有两个函数,write_rds() 和 read_rds():